-
实验室负责人
贺雄雷,中山大学教授、博士生导师,教育部长江学者特聘教授(2012年)。2007年从美国密歇根大学(University of Michigan-Ann Arbor)获博士学位,同年通过中山大学“百人计划”引进回国担任生命科学学院教授。长期从事遗传学和系统生物学研究。发现核小体通过抑制DNA双链的呼吸作用调节特定类型碱基突变的新机制;阐明了肿瘤细胞基因表达网络的演化动力和特征;揭示了腺苷琥珀酸裂解酶缺陷症的致病机理并提出一个有潜力的治疗方案。近年来聚焦个体发育过程中细胞群体的分化和演变。主持承担了国家自然科学基金重大研究计划集成项目、杰出青年基金项目、重点项目、科技部“973”课题、科技部万人计划科技创新领军人才项目等。作为通讯作者在包括Science,Nature Genetics,Nature Methods,Nature Communications,National Science Review,PNAS,Genome Research,MBE,JGG等学术期刊发表SCI论文30多篇,并受邀为Science撰写观点文章(Perspectives)。
-
主要研究方向
本实验室通过实验生物学与计算生物学相结合的方法,对基因组学、分子进化学与系统生物学相关领域问题进行研究。目前,实验室主要致力于以下几方面的研究:
1)基因与基因组演化的分子生物学研究:以酿酒酵母为研究对象,对突变的发生与调节、基因与基因组结构、基因互作网络的演化等分子进化学问题进行研究,对于基因型-表型网络的关系、加性遗传方差的起源与基因功能的进化学分析等方向具有独特的见解。
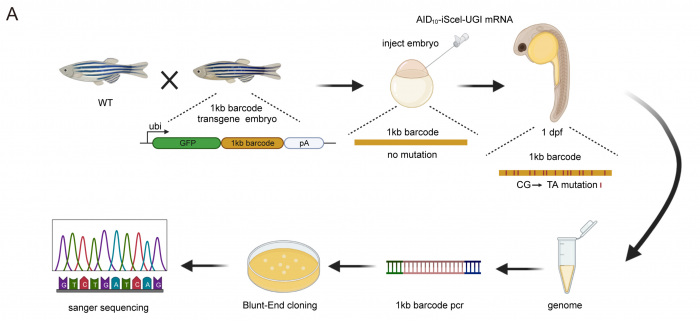
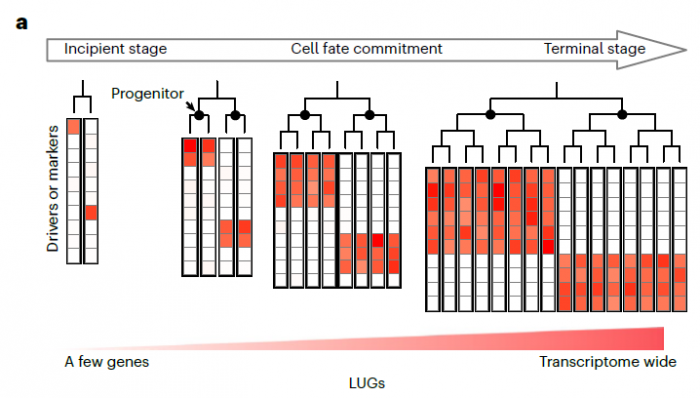
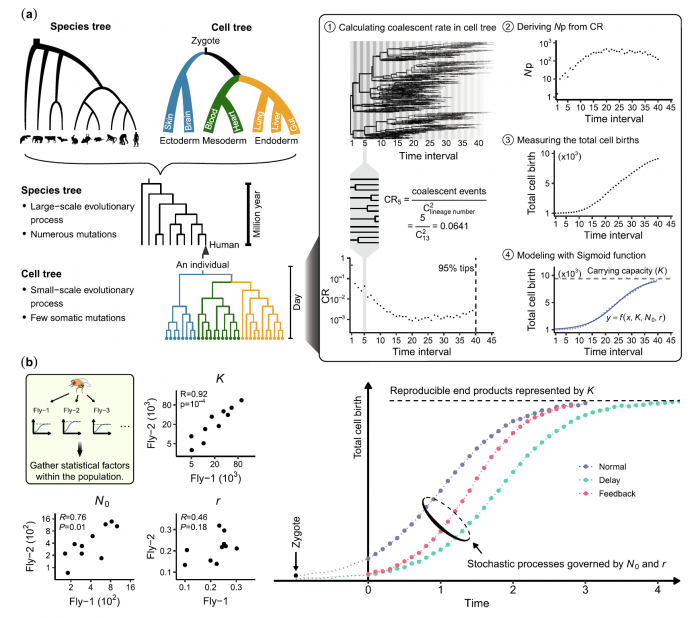
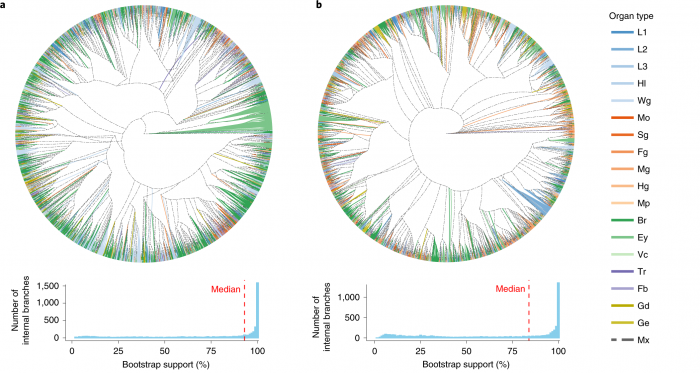
2)多细胞生物发育过程中细胞谱系研究:多细胞生物是从单个受精卵细胞经过细胞分裂与分化等过程逐步发育为成熟个体。利用本实验室自主开发的单细胞谱系追踪系统(substitution mutation-aided lineage-tracing system, SMALT),我们能够在果蝇、斑马鱼与小鼠等多个模式物种中,实现高分辨率的单细胞水平细胞谱系追踪。通过进一步整合细胞谱系信息与单细胞转录组等多组学数据,我们将构建分子动力学模型对多细胞生物的发育过程进行量化与描述。
3) 肿瘤发生、发展与演化的基因组学:从单个细胞发展到一个肿瘤的过程事实上就是一个短期的细胞演化事件,因此全程观察这一过程对了解肿瘤的发生、发展的机理非常有价值;不过,由于伦理等方面的原因我们通常只能获得具体一个或少数时间点的肿瘤样本,监测一个完整的肿瘤演化过程非常困难。基于此,我们建立了人体外的肿瘤演化模型,从基因组(CGH),外显子组,包括小RNA在内的转录组等不同层面探讨由正常细胞到肿瘤所发生的突变和选择的分子基础。